Grüne, pinke und türkise Faschingsschlangen tanzen auf dem Bildschirm von Gustav Oberdorfer und bilden immer neue Figuren. Aber der Schein trügt: Was aussieht wie fröhliche Deko, sind in Wahrheit komplexe Modelle von Aminosäuren, die sich in immer neuen Kombinationen ineinanderwinden. Oberdorfer arbeitet seit Februar 2018 als Universitätsassistent am Institut für Biochemie der TU Graz daran, mithilfe von Simulationssoftware Proteine zu designen. Was hier gerade passiert: Das Programm Rosetta berechnet die Faltung einer Aminosäurekette.
Proteine und ihre Faltung
Proteine bestehen aus aneinandergereihten Aminosäuren, den Aminosäureketten. Diese sind ineinandergefaltet und ergeben so die Struktur des jeweiligen Proteins, die häufig für dessen Funktion ausschlaggebend ist. Aminosäureketten falten sich dabei in die Struktur mit der geringsten freien Energie – doch um diese in der Forschung zu finden, müssen unzählige Varianten verschiedener Faltungen am Computer durchgespielt werden. „Müsste ein Protein alle möglichen Kombinationen während seiner Faltung ausprobieren, würde das länger dauern als die Zeitspanne, die unser Universum alt ist“, veranschaulicht Oberdorfer. Die Struktur eines Proteins vorherzusagen, geschweige denn zu designen, blieb aufgrund dieses sogenannten Faltungsproblems lang nur graue Theorie.
Doch in den letzten Jahren haben mehrere parallele Entwicklungen die Forschung radikal beschleunigt: „Es steht mehr Rechenleistung zur Verfügung, die Algorithmen der Simulationsprogramme werden immer besser und synthetische DNA-Fragmente zum Herstellen der künstlichen Proteine im Labor werden günstiger – sie können sogar mittels eines Verfahrens produziert werden, das ähnlich wie der Tintenstrahldruck funktioniert“, ist Oberdorfer begeistert. „Daher können wir heute synthetische Proteine in großem Maßstab testen. Forschende können nun etwa gezielt nach einem Protein suchen, das das Influenza-Virus bindet.“ So öffnet computergestütztes Proteindesign für viele biotechnologische und biomedizinische Probleme einen neuen Lösungsansatz.
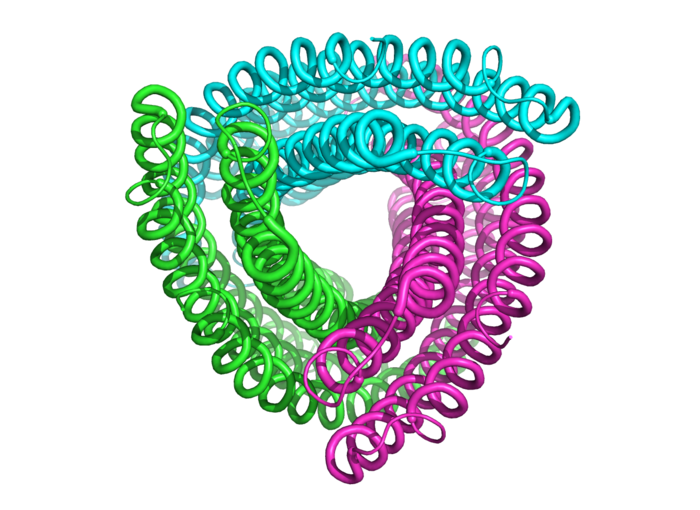
Keine Faschingsschlangen, sondern die Struktur einer Aminosäurekette.
Mit Aminosäureketten und deren Faltung beschäftigte sich Oberdorfer schon für seine Dissertation: Er war an einem Forschungsprojekt beteiligt, das untersuchte, wie Bakterien das Antibiotikum „Nikkomycin“ herstellen. Dies geschieht unter anderem mithilfe des Proteins „NikO“ – und die Struktur ebendieses Proteins bestimmte der Jungforscher. Seine heutige Arbeit sieht Oberdorfer als logische Fortsetzung: „Strukturbiologie und Proteindesign sind zwei Seiten derselben Münze.“
Filter und Ausbuchtungen
An der TU Graz leitet Gustav Oberdorfer derzeit zwei Forschungsprojekte: In einem FWF-Projekt werden in der Natur nicht vorkommende Proteinstrukturen gesucht, die in ihrer Mitte Hohlräume bilden. Diese können als Filter dienen und so im Praxiseinsatz beispielsweise toxische Verbindungen wie Quecksilber aus Wasser filtrieren.
Für sein zweites Forschungsprojekt wurde dem NAWI-Graz-Absolventen ein prestigeträchtiger ERC Starting Grant der Europäischen Kommission zugesprochen. Er will mit seinem Team Proteine mit Ausbuchtungen herstellen, die zum Beispiel ein kleines Molekül binden und in weiterer Folge auch eine Reaktion katalysieren können. „Wir möchten aber nicht nur einen Einzelfall lösen, sondern eine generelle Methode entwickeln, wie sich solche Proteine herstellen lassen“, präzisiert Oberdorfer. „Ob das funktioniert oder nicht, werden wir sehen: Vielleicht gibt es für diese Fragestellung gar keine Lösung, aber wenn wir es nicht versuchen, werden wir es nie wissen.“ Sein Team arbeitet mit konkreten Anwendungsbeispielen und wird etwa versuchen, das Pestizid Glyphosat zu binden und abzubauen.
Der Alltag des 36-Jährigen ist ein Wechselspiel aus Büroarbeit – beim Designen der Proteine am Computer – und Laborarbeit, bei der die künstlichen Proteine getestet werden. Letzteres ist für ihn übrigens der wichtigste Teil, „denn bis dahin ist alles nur ein theoretisches Konstrukt“.
Graz – Seattle – Graz
In Graz hat Gustav Oberdorfer, ein gebürtiger Steirer, auch schon studiert: Nach seinem Studium der Molekularbiologie an der Karl-Franzens-Universität absolvierte er das NAWI-Graz-Doktoratskolleg „Molekulare Enzymologie“ am Institut für Molekulare Biowissenschaften der Karl-Franzens-Universität und am Institut für Biochemie der TU Graz. Nach der Promotion zog es Oberdorfer in die Ferne, viereinhalb Jahre verbrachte er an der University of Washington in Seattle am Institute for Protein Design, einer der renommiertesten Forschungsstätten auf diesem Gebiet – die Universität war ihm bereits von einem sechsmonatigen Forschungsaufenthalt während seiner Dissertation bekannt.
„Die Zeit in den USA war die produktivste und interessanteste Phase in meinem bisherigen Forscherleben“, erzählt Oberdorfer. „Ich arbeitete dort mit über 80 Leuten in einem großen Labor, das war ein extrem kreatives Umfeld.“ Zwei Grundprinzipien förderten die gute Zusammenarbeit: „Erstens sind alle Ergebnisse für alle Forschenden zugänglich. Das heißt, dass man auch Erkenntnisse anderer übernehmen und daran weiterarbeiten kann – das ist etwas, das ich in dieser Form einzigartig gefunden habe. Wir nannten das ‚Leapfrogging‘, also Bockspringen – man lässt einzelne Stufen im Entwicklungsprozess aus, da man hier auf bestehende Ergebnisse zurückgreifen kann. Und zweitens wurde auf eine möglichst große Diversität der Arbeitsgruppe Wert gelegt – in fachlicher Hinsicht, aber etwa auch in kultureller.“

Gustav Oberdorfer im Labor, wo die künstlichen Enzyme getestet werden.
Auch wenn ihm nach dem bereichernden Auslandsaufenthalt die Rückkehr nach Österreich anfangs nicht leicht fiel, hat sich Oberdorfer mittlerweile wieder gut eingelebt: „Die Studierenden hier sind praxisbezogen besser ausgebildet, die Infrastruktur an den Universitäten ist top.“ An der TU Graz ist Oberdorfer auch in der Lehre tätig, hier hält er erstmals Vorlesungen. „Das war eine ganz neue Erfahrung für mich, nicht in einer Übung mit den Studierenden zusammenzuarbeiten, sondern Vorlesungen zu halten. Die Lehrveranstaltungen machen großen Spaß – ich arbeite gerne mit den Studierenden.“ Wer sich aus erster Hand über Oberdorfers Forschungsgebiet informieren möchte, kann schon bald seine neu konzipierte Vorlesung „Protein Design“ besuchen.
„Raus aufs Meer“
Neben der Arbeit gibt es für Gustav Oberdorfer noch drei weitere Fixpunkte im Leben: erstens den Bergsport, zweitens das Reisen – sei es bei einer dreiwöchigen Radtour durch Island oder beim Trekking in Nepal – und drittens die Musik, der Forscher spielt Bassgitarre in der Band „Musikcafe Prenner“. Hörtipp: Vergangenen Herbst wurde die neue EP „Raus aufs Meer“ vorgestellt.
Dieses Forschungsgebiet ist im FoE „Human & Biotechnology“ verankert, einem der fünf Stärkefelder der TU Graz. Mehr Forschungsnews aus diesen Bereichen finden Sie auf Planet Research.

